| 製品モデル | DNBSEQ-T20×2 | DNBSEQ-T10×4 | DNBSEQ-T7 | DNBSEQ-G400 | DNBSEQ-G99 | DNBSEQ-G50 |
| 特長 | 超高スループット | 超強力なカスタマイズ | 超高スループット | 融通性 | 効率性 | 効率性 |
| アプリケーション | 超高深度全ゲノムシーケンス | 大規模集団ゲノムシーケンシングプロジェクト | 大規模・中規模シーケンスプロジェクト | WGS、WES、トランスクリプトームシーケンスなど | ターゲット腫瘍学パネル、感染症、腫瘍学メチル化、小規模ゲノム | 小規模ゲノムシーケンス、ターゲットDNA・RNAシーケンス、低深度全ゲノムシーケンス |
| フローセルタイプ | スライド | FC | FC | FCL & FCS | FC | FCL&FCS |
| レーン/フローセル++ | 1レーン | 1レーン | 1レーン | 4レーンと2レーン | 1レーン | 1レーン |
| オペレーションモード | 超高スループット | 超高スループット | 超高スループット | 高スループット | 小、中スループット | 中スループット |
| 最大スループット/ラン | ~72Tb | 76.8Tb | 6Tb | 1440Gb | 48Gb | 150Gb |
| 有効リード数/フローセル | 35 B(PE100) | 45B(PE100)/32 B (PE150) | 5000M | 1500-1800M | 80M | 500M/100M |
| 平均ラン時間 | 60-80時間 | 96時間 | PE150、24~30時間 | FCS:17~37時間 FCL:14~109時間 | PE150、12時間 | 9~40時間 |
| 最小リード長 | PE100 | PE100 | PE100 | SE50 | SE100 | SE50 |
| 最大リード長 | PE150 | PE150 | PE150 | SE400/PE200 | PE150 | PE150 |
推奨アプリケーション
| アプリケーションタイプ | アプリケーション | サンプルタイプ | ライブラリ調製 | 適切なシーケンサー | シーケンスと解析 | レポート |
|---|---|---|---|---|---|---|
| 全ゲノムシーケンス | WGS | 血液、唾液、gDNA | DNBSEQ-G400、DNBSEQ-T7 | |||
| 全エクソームシーケンス | WES | 血液、唾液、gDNA | DNBSEQ-G400、DNBSEQ-T7 | |||
| 微生物検出 | 病原菌高速検出 | 血液、脳脊髄液、肺胞洗浄液 | DNBSEQ-G50 | |||
| 腸内微生物叢 | 糞便試料 | DNBSEQ-G50 | ||||
| 微生物検出と耐性分析 | 血液培養 | DNBSEQ-G50 | ||||
| 腫瘍 | WESトータルソリューション | FFPE、組織、血液 | DNBSEQ-G400 | |||
| RNA | トランスクリプトームシーケンス | 組織、細胞、gDNA、RNA | DNBSEQ-G400、DNBSEQ-T7 | |||
| RNAシーケンス | 組織、細胞、gDNA、RNA | DNBSEQ-G400、DNBSEQ-T7 |


MGIのDNAシーケンス装置は、DNBSEQ™と呼ばれる最先端のコアテクノロジーを採用しています。DNB(DNAナノボール)は流路に注入された後に、パターンアレイチップにロードされます。そしてシーケンスプライマーが添加され、DNBのアダプター領域にハイブリダイズします。蛍光標識されたdNTPプローブとDNAポリメラーゼを含むシーケンス試薬を注入すると、シーケンス反応が始まります。DNB上の蛍光標識されたプローブがレーザーにより励起された後に、画像を撮影します。その後、MGI独自のソフトウェアを使用して、画像をデジタル信号に変換します。この情報を使用してサンプルのDNA配列を決定します。
DNBSEQ™には、DNBに関連するすべてのシーケンステクノロジーが含まれています。具体的には、一本鎖環状化DNAからのDNB調製テクノロジー、パターンアレイ、DNBのローディング、cPAS(結合的プローブアンカー合成)テクノロジー、DNBのペアエンドシーケンス技術、流路系および画像撮影システム、ベースコールのアルゴリズムなどがあります。cPASテクノロジーは、BGISEQ-50、BGISEQ- 500、DNBSEQ-G50、DNBSEQ-G400、DNBSEQ-T7など、多様なシーケンスプラットフォームで幅広く使用されています。
他の既存シーケンスプラットフォームと比較すると、DNBSEQ™シーケンステクノロジーには「エラー蓄積の無いDNB調製」および「高密度パターンアレイ」の両方の特長があります。これらの特長によりシーケンス精度は劇的に改善され、WGS/WESアプリケーションにおいては重複率が大幅に低減されます。また、PCRフリーのライブラリ調製法と組み合わせれば、DNBSEQ™では他のプラットフォームよりも優れたSNPおよびIndel検出精度を実現できます。さらに、index hoppingの割合も、DNBSEQ™は他のプラットフォームを大きく下回っています。
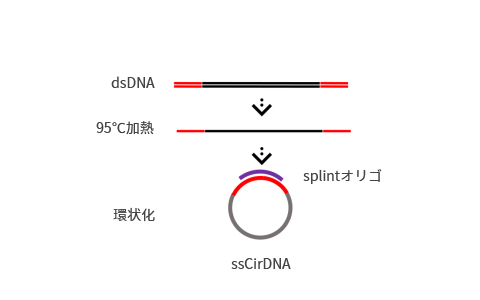
シーケンスチップへのロード前に、DNB濃度はQubit測定により簡単に定量することができます。高額な定量化装置や試薬は必要ありません。ローリングサークル増幅(RCA)の主なメリットとしては、増副におけるエラー発生率の減少が挙げられます。RCAには忠実度が極めて高いDNAポリメラーゼを使用し、サイクルごとにオリジナルコピーがテンプレートとして使用されます。これにより、DNBが100~1、000コピーに複製される間に、同じ位置で増幅エラーが蓄積することをを回避することができます。さらに、RCAテクノロジーではPCRなどの他の増幅手法で認められるような、エラー、GCバイアス、ドロップアウトの指数関数的な蓄積を避けることができます。これらすべてが、DNBSEQプラットフォームにおけるシーケンス精度の大幅な向上を実現します。
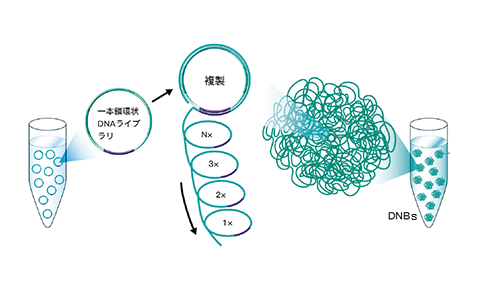
DNB は生成の過程は、スライド表面の結合部位と同じサイズになるように最適化されています。そのため、各結合部位には DNB が1 つしかロードされず、フローセル面積を有効に活用することができます。

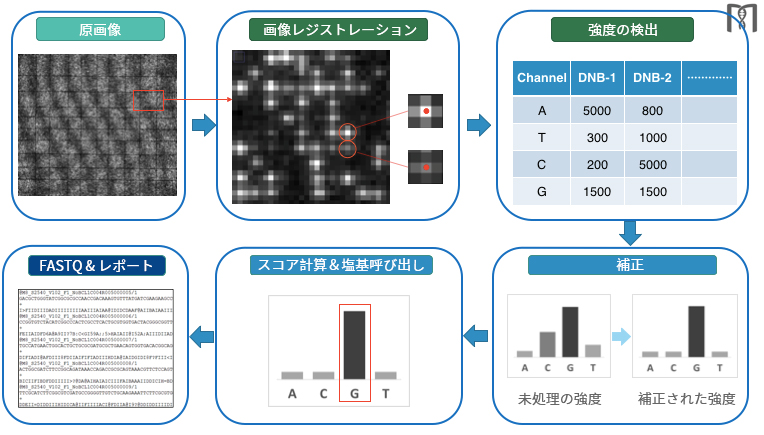
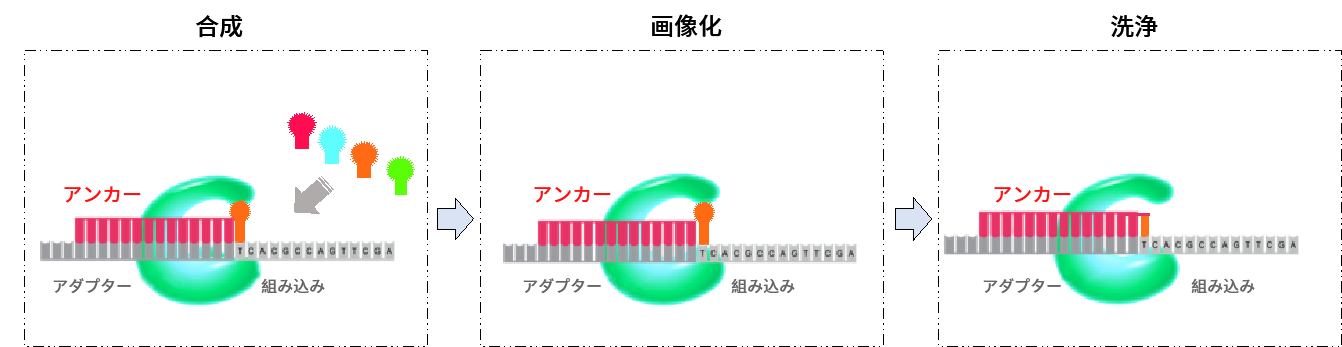
cPAS 技術:シーケンシングプライマーを DNB のアダプター領域とハイブリダイズさせた後、DNA ポリメラーゼを用いて蛍光標識 dNTP プローブを組み込みます。次に、結合されていない dNTP プローブを洗い流し、DNB フローセルをスキャンします(図 :イメージング)。蛍光シグナルをデジタル信号に変換し、MGI 独自のベースコールソフトウェアを使用して塩基配列決定します。イメージング後は再生試薬を加えて蛍光色素を除去し、DNBを次のサイクルに備えます。
数万の突然変異体からスクリーニングされた優れたシーケンシング用ポリメラーゼの採用によってシーケンスケミストリーは大いに進歩しており、1塩基の反応時間は 1 分未満に短縮されています。
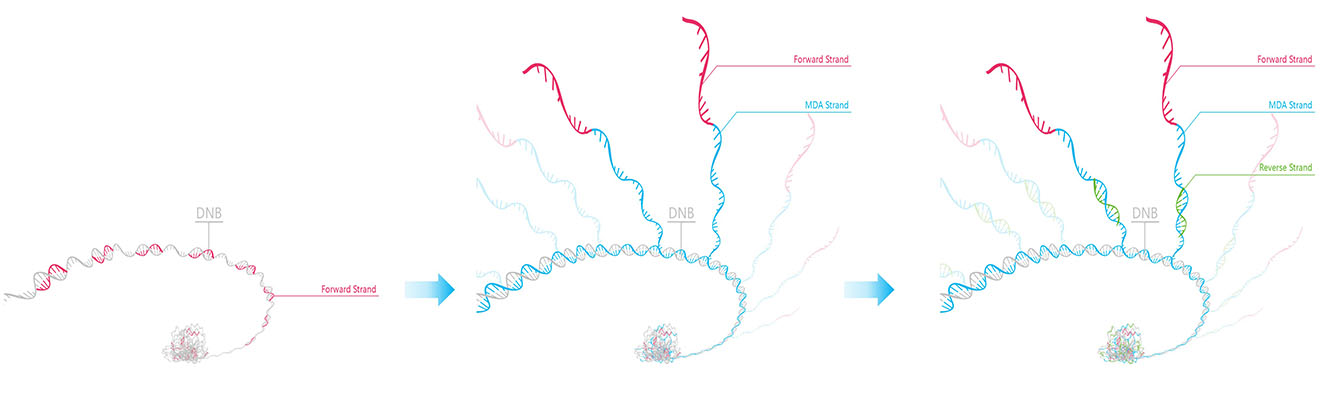
1st ストランドのシーケンシングが完了したら、相補鎖合成用のプライマーおよび鎖置換型ポリメラーゼを添加して、2nd ストランドの合成を開始します。新たに添加したプライマーからを伸長した鎖が、すでに塩基配列が決定されたオリジナルの鎖に到達すると、そこからオリジナルのシーケンシング鎖を置換しながら新しい鎖を合成します。この新たに生成した鎖は(2nd ストランド)オリジナルの DNB に結合した状態が保たれつつも、鎖長が最大になるように最適化されています。この2nd ストランドにシーケンシングプライマーをハイブリダイズさせた後、同じケミストリーによりシーケンシングを行います。2nd ストランド鎖にはより多くのインサート DNA のコピーが含まれており、1st ストランドよりもはるかに強いシグナルが得られ、シーケンシングの精度が向上します。
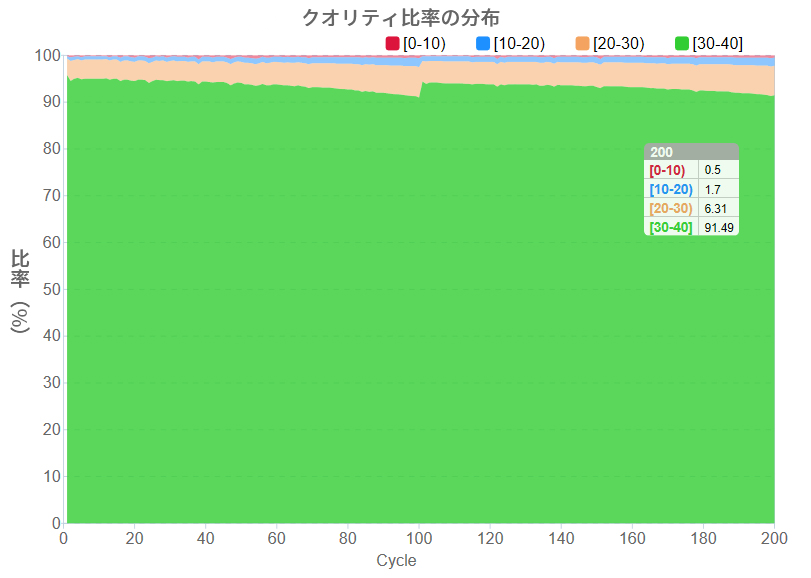
ベースコールおよびベースコールのクオリティは、すべてのチャネルのシグナル強度に基づいて算出されます。シグナルの特性評価とシーケンシングエラーの関係は、既知のデータモデルに基づいて確立されています。未知のサンプルについての予測シーケンシングエラーは、シグナルの特性評価に基づいて算出されます。クオリティスコアは、phred-33 基準に基づいて算出されます。
MGI は、独自のサブピクセル画像レジストレーションアルゴリズムを開発しました。これにより、サブピクセルレベルで画像の強度を検出することができ、またベースコールの精度が大幅に向上します。
当社は業界トップクラスのテクノロジーを駆使し、GPU アクセラレーションアルゴリズムを組み入れて、実行効率を最適化するとともに、リアルタイムの画像解析およびベースコールを採用することで、データ処理の速度と精度を飛躍的に向上させました。